Bazy danych o białkach
Znajomość trójwymiarowej struktury trzeciorzędowej białka jest kluczowa dla zrozumienia jego funkcji biologicznej. Określenie sekwencji w białku jest obecnie zadaniem rutynowym, natomiast określenie struktury jest dużo trudniejsze. W 1992 r. znano strukturę atomową około 400 białek, 10 lat później — ponad 14 500. Zdecydowaną większość z nich określono w wyniku otrzymania białek w postaci krystalicznej. W pewnych warunkach możliwe jest wytworzenie kryształu białka z jego wodnego roztworu (w węzłach sieci takiego kryształu znajdują się molekuły białka) i przeprowadzenie strukturalnej analizy rentgenowskiej. Druga co do znaczenia metoda wyznaczania struktury to technika jądrowego rezonansu magnetycznego, która nie wymaga uprzedniej krystalizacji. W ten sposób wyznaczono ok. 17% znanych struktur. Około 2% struktur wyznaczono na drodze modelowania teoretycznego.
Struktury białek deponowane są w komputerowych bazach danych. Istnieją niepubliczne i publicznie dostępne bazy danych. Te pierwsze prowadzone są na ogół przez firmy farmaceutyczne. Z tych drugich najbardziej znana jest internetowa baza danych o nazwie Protein Data Bank (PDB) i o adresie www.rcsb.org/pdb/, gdzie rcsb jest skrótem od The Research Collaboratory for Structural Bioinformatics. PDB jest konsorcjum, w którym uczestniczą cztery amerykańskie instytucje: Rutgers University w New Jersey, University of California w San Diego, San Diego Supercomputer Center, i National Institute of Standards and Technology w Maryland. Dane zgromadzone w PDB określają przede wszystkim współrzędne atomów w strukturze natywnej białka, w tym w szczególności współrzędne Cα. W PDB zawarte są też informacje o strukturach drugorzędowych, które można uzyskać na dwa sposoby. Po pierwsze określenie struktur drugorzędowych dokonywane jest przez autorów deponujących dane, po drugie odbywa się poprzez analizę przeprowadzaną automatycznie przez PDB. Te dwa rodzaje danych o strukturze nie są ze sobą w pełni zgodne. Rozbieżności dotyczą najczęściej granic struktur drugorzędowych. Oprócz danych o strukturze, PDB udostępnia również program graficzny RASMOL, który pozwala wykreślić dane strukturalne.

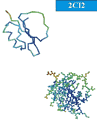
Rysunek 12. Schematyczna reprezentacja białka 2CI2 uzyskana z Protein Data Bank
(aby obejrzeć powiększony rysunek, kliknij w miniaturkę)
Jako przykład zaznajomienia się ze strukturą białka rozpatrzmy białko zwane inhibitorem trypsyny. Ma ono w PDB kod 2CI2 i jest zbudowane z 83 aminokwasów. Strukturę trzeciorzędową ostatnich 65 z nich udało się określić drogą dyfrakcji rentgenowskiej. 13 aminokwasów (od aminokwasu 31 do 43 według deponujących dane) tworzy α-helisę. Istnieją też cztery nici β, z których dwie są długie: aminokwasy 47 - 53 i 64 - 71, a dwie krótkie: aminokwasy 22 - 24 i 80 - 83. Wszystkie cztery nici tworzą w sumie jedną strukturę β. PDB udostępnia schematyczny wizerunek struktury przedstawiony na rys. 12. Najkrótsza nić β nie jest w tej reprezentacji obecna. Jest to przykład różnic w sposobie klasyfikowania i przedstawiania struktur białek dostępnych poprzez PDB i otrzymywanych doświadczalnie. Zdeponowane w internetowych bankach danych struktury białek można też analizować samodzielnie.
Rysunek 13. Dwa rysunki ilustrujące budowę białka 2CI2. Górny rysunek przedstawia kształt szkieletu, a dolny przedstawia położenia atomów i schemat utworzonych wiązań
(aby obejrzeć powiększony rysunek, kliknij w miniaturkę)
Rys. 13 przedstawia reprezentacje szkieletu (górny panel) i całej struktury atomowej białka 2CI2 (dolny panel) wykonane w programie graficznym RASMOL. W konwencji RASMOL-u rozmiary wszystkich atomów są jednakowe i zostały tu dobrane tak, żeby widoczne były połączenia między atomami. Struktury drugorzędowe widać na rysunku samego szkieletu, ale byłoby bardzo trudno je dostrzec, gdyby wykreślić również łańcuchy boczne. W przypadku użycia promieni atomowych van der Waalsa (odpowiadających rozmiarom twardego rdzenia atomu i zgodnych z równaniem van der Waalsa dla gazu), wizerunek białka wyglądałby jak zwarta globula zbudowana z gęsto upakowanych kul. Globula ta miałaby szorstką powierzchnię. Tak więc istnieje wiele technik przedstawiania wizerunków białek. Wszystkie one są dosyć umowne, gdyż dotyczą świata fizyki kwantowej. Należy tu jednak zaznaczyć, że wiele właściwości białek, np. takich jak ich ruch, jest już praktycznie w obszarze fizyki klasycznej z uwagi na stosunkowo duże masy molekuł.

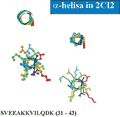
Rysunek 14. Cztery reprezentacje α-helisy w białku 2CI2 widzianej z profilu. Kolejno od góry do dołu są to: schemat wstążkowy szkieletu, wiązania tworzące szkielet, położenia atomów i schemat wiązań między nimi oraz schemat wszystkich wiązań chemicznych
(aby obejrzeć powiększony rysunek, kliknij w miniaturkę)
Przyjrzyjmy się teraz fragmentom struktury białka 2CI2. Rys. 14 przedstawia różne reprezentacje α-helisy jeśli patrzeć na nią "z boku", a rys. 15 dotyczy jej obrazu widzianego wzdłuż osi. Rysunek w kształcie zwiniętej wstążki jest schematyczny. "Kanciasty" rysunek szkieletu powstaje wtedy, gdy połączyć odcinkami położenia atomów węgli Cα. Kolejne panele uwzględniają łańcuchy boczne — wraz z wizerunkami atomów i bez. Symbole literowe określają kolejne aminokwasy, które występują w strukturze.
Rysunek 15. Tak jak na rys. 14, ale dla α-helisy widzianej wzdłuż jej osi
(aby obejrzeć powiększony rysunek, kliknij w miniaturkę)
Rys. 16 przedstawia różne reprezentacje dwóch dłuższych nici β, które są do siebie równoległe. Wreszcie rys. 17 ilustruje trzecią strukturę drugorzędową — skręt. Struktura ta dotyczy aminokwasów 62 - 65.

Rysunek 16. Trzy reprezentacje dwóch nici β w białku 2CI2
(aby obejrzeć powiększony rysunek, kliknij w miniaturkę)

Rysunek 17. Trzy reprezentacje skrętu w białku 2CI2 dla aminokwasów w pozycjach 62 - 65
(aby obejrzeć powiększony rysunek, kliknij w miniaturkę)
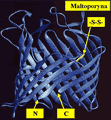
Białko 2CI2 jest globularne. Jeśli chodzi o białka membranowe, to w sierpniu 2003 r. PDB miał w swoich zbiorach tylko 67 takich białek. Białek membranowych jest w przyrodzie mniej więcej tyle, co globularnych, ale można je wyizolować tylko w specjalnie wybranych detergentach. Z punktu widzenia przemysłu farmaceutycznego są one ważniejsze niż białka globularne, gdyż aż 2/3 obiektów działania lekarstw na poziomie molekularnym to białka membranowe. Rys. 18 przedstawia schematyczną reprezentację białka membranowego, zwanego maltoporyną, uzyskaną ze szwajcarskiej bazy danych SWISSPROT, o adresie www.expasy.ch/sprot/. Białko to wygląda jak rurkowy kanał. Jest ono zbudowane z wielu nici β, które są spięte mostkiem dwusiarczkowym zaznaczonym na żółto.